Alex N. Nguyen Ba 等人在《Nature》的一篇名为 “High-resolution lineage tracking reveals traveling wave of adaptation in laboratory yeast” 的论文种所报告的实验。
救命啊,有人转基因啦~
隔壁组是做演化与生态学的,暑假有个本科生来做项目。这么短的时间只能拿现有的数据做文章,这个数据来自 Alex N. Nguyen Ba 等人在《Nature》的一篇名为 "High-resolution lineage tracking reveals traveling wave of adaptation in laboratory yeast" 的论文。
论文所报告的实验简而言之:
- 在初始时刻给每一个酵母的基因组插入一个独特的 DNA 序列(也就是所谓的条形码,繁殖后拥有同一个条形码的酵母群被认为是同一个“物种”(species));
- 让这群酵母繁殖 100 代,在此期间根据监控拥有不同条形码的酵母在整个群体中所占比例及其变化,及其对环境的适应度 (fitness);
- 100 代后在原有的条形码旁边再插入新的各自不同的 DNA 序列,(因为之前的条形码经过演化后,很多被挤兑到比例可以忽略不计,另外一些占据的比例过大,其中的后代可能发生了新的分化);
- 跳转到第 2 步……
条形码的插入
条形码的插入利用的是酶催化的基因重组。整个系统叫做 Cre-Lox
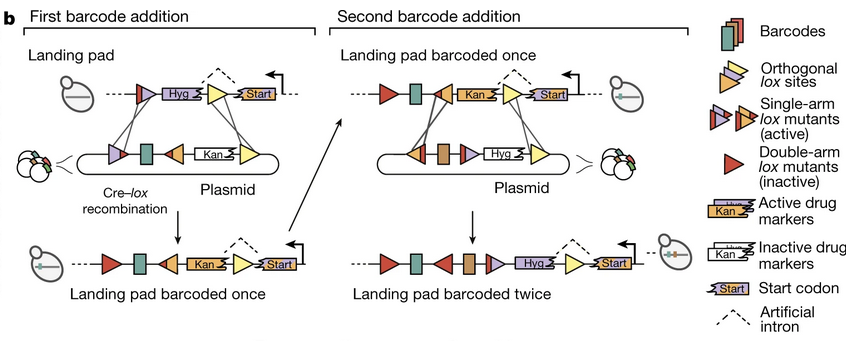
Lox 是一段基因,有多种不同的类型。每一种都是中间有一段名为 spacer 的内容不定的序列,spacer 两侧的内容确定的序列被称作臂 (arm);臂的不同具体序列定义了不同的 Lox 类型。论文的示意图中用三角形表示。
Cre 是一种重组酶 (recombinease),在它存在的环境里,距离接近的、具有同种 Lox 基因的两条 DNA 有一定概率从 Lox 位点断开,然后交换半截之后重新连起来。这种交换重连叫做基因重组。
这个实验的对象是一种常见的出芽酵母 (budding yeast, Saccharomyces cerevisiae) 的二倍体,其 HO 基因的位置已经被修改过,变成了启动子、Cre 基因、两个不同的 Lox 片段、两 Lox 中间夹着一段选育标记 (selection marker)。
条形码位于外源的质粒 (plasmid) 上,通过 transformation 送入酵母细胞内。条形码基因两侧紧邻着两个 Lox 片段,右侧有一个和酵母体内不同的选育标记,选育标记的右侧还有第三个 Lox 位点。
Cre-Lox 系统的基因重组一般是可逆反应,不利于插入条形码的效率。为了解决这个问题,作者发现 Lox 的两臂可以发生基因突变,当只有一臂突变时对重组的影响很小,两臂都突变后的 Lox 就几乎不会在 Cre 催化下重组了。
所以酵母基因组和质粒最左侧的 Lox 各有不同的一臂是突变型,条形码插入后两臂都成了突变型,就不会再通过重组离开酵母的基因组;
条形码右侧紧邻的 Lox 还是单臂突变型,这就是下次条形码插入的起始位置。利用同样的原理,可以多次插入不同的条形码。
插入完成后,我们还希望质粒被细胞排除掉。这一点通过将酵母细胞置于 5-氟乳清酸 (5-FOA) 环境中实现。因为质粒上带有 Ura3 基因作为选育标记,可以自主合成尿嘧啶 (Uracil),两者反应生成有毒的 5-氟尿嘧啶 (Fluorouracil),所以能在筛选环境中存活的酵母都已经不含质粒。
细胞培养、数据读取和分析
培养时,5 毫升酵母细胞悬液被放在 15 毫升玻璃试管里,在 30 °C 环境保持摇匀。据之前论文说,这种条件下细胞每天增殖 10 代。作者每天将细胞悬液稀释 倍,并取一定体积的悬液进行基因测序;每 10 天进行一次新的条形码标记,称为 1 个 epoch;实验一共进行了 10 个 epochs。
基因测序的目的是,在某一时刻,数出携带每种条形码的酵母的数目。因为酵母是单细胞生物,每个细胞内只有一个条形码,也就是数出各种条形码的分子数。
方法是在条形码的上下游选择引物位点,对 DNA 分子做 PCR 扩增,让 DNA 分子的数量足够显著,根据 PCR 的循环数倒推原来的分子数。
为了让 PCR 过程本身不给实验引入额外的误差,给每个 DNA 添加了一段唯一的分子标记 (UMI)。方法是将 PCR 本身分成两个阶段,第一阶段 PCR 的 5' 端引物有一段 8-12 个碱基的随机序列;第二阶段的 PCR 按正常配置进行。最终拥有同一分子标记的 PCR 产物可以看作最初来自同一个条形码分子。
比较短的基因序列,可以直接交给生物科技公司(本文是 Illumina) 读取序列信息,时间和财务成本都可负担。
读取到序列信息之后,作者自己用 Python 的 regex 库写了基于正则表达式的算法来进行分析。
而演化的核心概念适应度 (fitness),在本实验中的测量方法是:把某个条形码的一群酵母,和等量的添加了荧光标记的基准酵母放在一起生长,经过相同的时间之后,观察有荧光的细胞在全体细胞中所占的比例,据此测量这个条形码代表的酵母的对基准酵母的排挤能力。这种方法叫做 "competitive fitness assays"。
酵母的性别
这段对理解文章没什么用,但是反正已经写了……
酵母是一种单细胞真核生物,可以进行有性生殖,自然环境中一般是二倍体。进行有性生殖时,二倍体细胞先减数分裂成单倍体细胞,单倍体有两种可能的交配型 MATα 和 MATa,两者的区别就是 MAT 基因的内容是 α 还是 a;两种交配型的单倍体融合成新的二倍体,就完成了有性生殖。
两种交配型都有一个名为 HO 的基因,编码一种内切酶。
在天然环境中,HO 可以在 MAT 位点造成 DNA 双链断裂,引发同源重组,从而将交配型从 MATa 切换为 MATα,或反之,从而改变单倍型的性别。这样酵母就可以在不突变的情况下通过有性生殖增加基因多样性。
而在实验室中,科研人员更希望酵母不要自行改变性别,因此经常把 HO 基因敲除,或利用 HO 位点作为基因工程的插入位点,比如本实验中条形码插入的位置。Figure 1(b) 左上角表示的这段基因就是从 HO 位置修改而来的。
本文收录于以下合集: